時間序列分析是根據系統觀測得到的時間序列數據,通過曲線擬合和參數估計來建立數學模型的理論和方法。時間序列分析常用在企業經營管理、市場潛量預測、農作物病蟲災害預報、環境污染控制、生態平衡、天文學和海洋學等方面。
今天來針對台灣的護國神山股價的走勢作預測分析,雖然購買股票聽起來很有誘惑力,但我們更應該進行詳細的深入分析,避免以投機為基礎的股票購買。
1.載入相關套件
library(PerformanceAnalytics)
library(quantmod)
library(stringr)
library(forecast)
2.取得TSMC自 2020/1/1 開始的股價 (yahoo的台股股價名稱要用代號.TW)
getSymbols("2330.TW", src="yahoo", from="2020-01-01")
TSMC.TW <- get("2330.TW")
將第四欄位(收盤價)存在變數TSMC_close_price
TSMC_close_price <- TSMC.TW[, 4]
fit.a <- auto.arima(TSMC_close_price, seasonal=F)
可以先看一下時間序列分析圖
tsdisplay(residuals(fit.a), lag.max=40, main="(3,1,4 Model Residuals)")
最上圖是時間序列的趨勢圖,判斷趨勢是否平穩,
下圖左ACF是畫自相關圖,右邊PACF是畫偏相關圖,用來判斷拖尾和截尾。
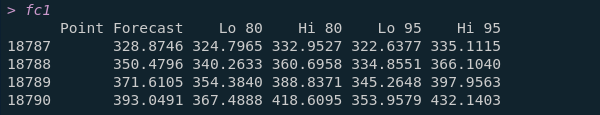
4.對 fit.a 做10天的預測(forecast),結果存於 fc1
fc1 <- forecast(fit.a, h=7)
畫出預測趨勢圖
plot(fc1)
fc1的預測低值/高值 可以輸入fc1$lower 或 fc1$upper 查看:

ACF和PACF概念:
ACF(自相關系數)描述的是現在股價與過去某時期之間的所有價格是否相關。PACF(偏自相關系數)描述的是現在的價格單純地和過去某一個價格的相關關系。兩者均為取值範圍 -1~1,絕對值越靠近1,說明相關關系越明顯;絕對值越靠近0,說明兩者之間線性相關很弱。藍色虛線表示的是誤差範圍,當值沒有超過藍色虛線的時候,基本可以認為他們不相關。如:
par(mfrow = c(1, 2))
acf(TSMC.TW$`2330.TW.Close`, main="Series tsmc")
pacf(TSMC.TW$`2330.TW.Close`, main="Series tsmc")
auto.arima(TSMC_close_price, seasonal = F)
對於 MA 模型,我們將使用 ACF 圖來識別 (q) 階,並且 PACF 將呈指數衰減。
如果我們查看 PACF 圖,我們可以注意到它僅在第一次滯後時有一個顯著的尖峰,
這意味著所有高階自相關都可以通過第一次滯後自相關有效地解釋。
如果要尋找最優模型:
先列出 AUTO ARIMA模型:
auto.arima(TSMC_close_price, trace=T)
data_d1=diff(TSMC_close_price, 1) #進行一階差分
tsdisplay(data_d1) #進行一階差分得到acf和pacf
data_d2=diff(TSMC_close_price, 2) #進行二階差分
tsdisplay(data_d2) #得到acf和pacf
由上圖可以看出二階差分的PACF有明顯 spike,由於我們使用 AUTO ARIMA 函數為我們提供了更好的數據集方法,因此我們就可以不用再花時間去找模型參數。
我們可以利用這個方式來檢驗過去的資料,例如在2021年5月份台股經歷一次大跌,TSMC股價也是無法倖免,在5/13最低價$547,我們可以觀測TSMC 2020/1/1到2021/5/2股價,然後預測2021/5/3~2021/6/11 的股價(30天),驗證是否落在真實的股價區間。
將觀測區間股價轉成 ts 時間序列(time series)
tsmc.ts <- ts(TSMC_close_price)
時間序列長度就是觀測區間的天數(348天),存於test.end.ts
(test.end.ts <- length(TSMC_close_price))
將天數往前30天(第318天)
(test.start.ts <- end.ts - 30)
測試資料從318~348天
stock_test<- window(tsmc.ts, start=test.start.ts, end=test.end.ts)
訓練模型從第一天(筆)資料至317天
stock_train <- window(tsmc.ts, start=1, end=(test.start.ts - 1))
對 stock_train 做auto arima
(fit <- auto.arima(stock_train))
然後forecast 30天的資料
fc <- forecast(fit, h=30)
使用自動擬合模型並檢驗殘差
checkresiduals(fit)
繪圖
plot(fc)
將預測期間的股價用紅色線段繪出
lines(stock_test, col="red")
利用ARIMA(0,1,1) 模型預測30個值(藍線),與之前我們預留的30個觀測值(紅線)進行對比,如下圖:
我們可以看出在5月份股價雖然跌至547,仍然在我們做的預測模型區間內,我們可以看forecast的upper與lower值,最低預測在539:
我們發現雖然模型從統計學意義上來說是擬合的。預測表明TSMC股價應該呈現上漲趨勢,但是由於突發事件的影響,預測值與觀測值的趨勢並沒有很好地重合,但是區間是吻合的。